Packages
Contents
tidyverseの関数群を使って心理学のデータの処理をするとして、各個人の尺度得点をそれぞれ出したい場合はおそらくdplyr::rowwise()とそれに関連する関数を使って処理すると思います。今回は別のアプローチでトライしてみました。
demo data
psych::bfiがちょうどいいので、逆転処理などをしておいてデモデータにします。
Code
df_bfi <-
psych::bfi |>
relocate(
gender, education, age
) |>
rownames_to_column(var = "id") |>
mutate(
across(
.cols = all_of(
unlist(bfi.keys) |>
str_subset(pattern = "^-") |> # extract reverse item
str_remove(pattern = "^-")
),
.fns = \(x) {7 - x} # six point scale, so subtract from seven.
)
) |>
1 rename(
q_education = education,
q_age = age,
q_gender = gender
) |>
as_tibble() # for better printing- 1
-
わざわざ列名を変えなくてもいいのですが、
tidyselect::starts_with()でいちいちignore.case = TRUEとしないとageとA*、educationとE*の区別をつけてもらえなくてめんどくさいので、変えておきます。
# A tibble: 2,800 × 29
id q_gender q_education q_age A1 A2 A3 A4 A5 C1 C2 C3 C4 C5 E1 E2
<chr> <int> <int> <int> <dbl> <int> <int> <int> <int> <int> <int> <int> <dbl> <dbl> <dbl> <dbl>
1 61617 1 NA 16 5 4 3 4 4 2 3 3 3 3 4 4
2 61618 2 NA 18 5 4 5 2 5 5 4 4 4 3 6 6
3 61620 2 NA 17 2 4 5 4 4 4 5 4 5 2 5 3
4 61621 2 NA 17 3 4 6 5 5 4 4 3 2 2 2 4
5 61622 1 NA 17 5 3 3 4 5 4 4 5 4 5 5 5
6 61623 2 3 21 1 6 5 6 5 6 6 6 6 4 5 6
7 61624 1 NA 18 5 5 5 3 5 5 4 4 5 4 3 4
8 61629 1 2 19 3 3 1 5 1 3 2 4 5 3 4 1
9 61630 1 1 19 3 3 6 3 3 6 6 3 3 2 2 4
10 61633 2 NA 17 5 5 6 6 5 6 5 6 5 6 5 5
# ℹ 2,790 more rows
# ℹ 13 more variables: E3 <int>, E4 <int>, E5 <int>, N1 <int>, N2 <int>, N3 <int>, N4 <int>, N5 <int>,
# O1 <int>, O2 <dbl>, O3 <int>, O4 <int>, O5 <dbl>rowwise()
心理学のデータ処理で因子分析とα係数の確認を終えたら、次に行うのは尺度得点の計算だと思います。参加者ごとに下位尺度の得点を算出していくのですが、その場合は調べてみるとたいていはdplyr::rowise()1とdplyr::c_across()2を使った処理に行き着くと思います。つまり、rowwise()で行ごとにグルーピングして、c_acrossでまとめたい要素をまとめて、それで処理するという方法です。
df_bfi |>
rowwise() |>
mutate(
score_A = mean(c_across(starts_with("A"))),
score_C = mean(c_across(starts_with("C"))),
score_E = mean(c_across(starts_with("E"))),
score_N = mean(c_across(starts_with("N"))),
score_O = mean(c_across(starts_with("O"))),
.after = q_age
) |>
ungroup()# A tibble: 2,800 × 34
id q_gender q_education q_age score_A score_C score_E score_N score_O A1 A2 A3 A4 A5
<chr> <int> <int> <int> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <int> <int> <int> <int>
1 61617 1 NA 16 4 2.8 3.8 2.8 3 5 4 3 4 4
2 61618 2 NA 18 4.2 4 5 3.8 4 5 4 5 2 5
3 61620 2 NA 17 3.8 4 4.2 3.6 4.8 2 4 5 4 4
4 61621 2 NA 17 4.6 3 3.6 2.8 3.2 3 4 6 5 5
5 61622 1 NA 17 4 4.4 4.8 3.2 3.6 5 3 3 4 5
6 61623 2 3 21 4.6 5.6 5.6 3 5 1 6 5 6 5
7 61624 1 NA 18 4.6 4.4 4.2 1.4 5.4 5 5 5 3 5
8 61629 1 2 19 2.6 3.4 2.4 4.2 4.2 3 3 1 5 1
9 61630 1 1 19 3.6 4 NA 3.6 5 3 3 6 3 3
10 61633 2 NA 17 5.4 5.6 4.8 4.2 5.2 5 5 6 6 5
# ℹ 2,790 more rows
# ℹ 20 more variables: C1 <int>, C2 <int>, C3 <int>, C4 <dbl>, C5 <dbl>, E1 <dbl>, E2 <dbl>, E3 <int>,
# E4 <int>, E5 <int>, N1 <int>, N2 <int>, N3 <int>, N4 <int>, N5 <int>, O1 <int>, O2 <dbl>, O3 <int>,
# O4 <int>, O5 <dbl>これで処理できるのでそれはそれでいいのですが、いくつか気になる点があります。
dplyr::ungroup()が必要ungroup()を忘れるとrowwiseグループが維持されるので、その後の処理で面倒なことが起こる可能性があります。行数によっては遅い
行数が少なければ気にならないと思いますが、行数が多いデータだと
rowwise()処理は体感できるレベルで遅いです。今回の2800行のデータだと4秒程度かかります。
microbenchmark::microbenchmark(
"rowwise" = {
df_bfi |>
rowwise() |>
mutate(
score_A = mean(c_across(starts_with("A"))),
score_C = mean(c_across(starts_with("C"))),
score_E = mean(c_across(starts_with("E"))),
score_N = mean(c_across(starts_with("N"))),
score_O = mean(c_across(starts_with("O"))),
.after = q_age
) |>
ungroup()
},
1 times = 5L
)- 1
- レンダリングの際にこの処理だけであまりにも時間がかかるので、ベンチマークの反復はデフォルトの100回から5回に減らしました。
Unit: seconds
expr min lq mean median uq max neval
rowwise 4.379653 4.452662 4.606178 4.665492 4.692358 4.840726 5というわけで、別のやり方がないか模索したわけです。
.by argument
グルーピングに.by3引数を用いるやり方です。dplyr::group_by()やrowwiseは関数としてパイプフローに組み込んで、処理の後もグルーピングを維持するのに対して.by = .../by = ...は処理の関数の引数で設定し、その処理限りのグルーピングを行います。戻ってくるdataframeはグループ化されていないので、個人的にはその後の処理がやりやすい感じがしてよく使っています。変数選択にはtidy-selectの文法が使えます。
なお、.by引数に突っ込めるrowwise()ってある?という質問がPosit Communityのforumに投げられているのですが、悲しいことに回答なしでclosedになっています。おそらく現時点ではそのようなものは実装されてないみたいなので、質問者の方が提示しているように.byには一意のID列を設定すればいいのだと思います。 そして、このID列を.byに入れたときは実質rowwise処理になるので、なんとc_across()がちゃんと動きます。
df_bfi |>
mutate(
score_A = mean(c_across(starts_with("A"))),
score_C = mean(c_across(starts_with("C"))),
score_E = mean(c_across(starts_with("E"))),
score_N = mean(c_across(starts_with("N"))),
score_O = mean(c_across(starts_with("O"))),
.after = q_age,
.by = id
)# A tibble: 2,800 × 34
id q_gender q_education q_age score_A score_C score_E score_N score_O A1 A2 A3 A4 A5
<chr> <int> <int> <int> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <int> <int> <int> <int>
1 61617 1 NA 16 4 2.8 3.8 2.8 3 5 4 3 4 4
2 61618 2 NA 18 4.2 4 5 3.8 4 5 4 5 2 5
3 61620 2 NA 17 3.8 4 4.2 3.6 4.8 2 4 5 4 4
4 61621 2 NA 17 4.6 3 3.6 2.8 3.2 3 4 6 5 5
5 61622 1 NA 17 4 4.4 4.8 3.2 3.6 5 3 3 4 5
6 61623 2 3 21 4.6 5.6 5.6 3 5 1 6 5 6 5
7 61624 1 NA 18 4.6 4.4 4.2 1.4 5.4 5 5 5 3 5
8 61629 1 2 19 2.6 3.4 2.4 4.2 4.2 3 3 1 5 1
9 61630 1 1 19 3.6 4 NA 3.6 5 3 3 6 3 3
10 61633 2 NA 17 5.4 5.6 4.8 4.2 5.2 5 5 6 6 5
# ℹ 2,790 more rows
# ℹ 20 more variables: C1 <int>, C2 <int>, C3 <int>, C4 <dbl>, C5 <dbl>, E1 <dbl>, E2 <dbl>, E3 <int>,
# E4 <int>, E5 <int>, N1 <int>, N2 <int>, N3 <int>, N4 <int>, N5 <int>, O1 <int>, O2 <dbl>, O3 <int>,
# O4 <int>, O5 <dbl>先ほどのrowwise()の書き方と全く同じ結果が返ってきています。ID列をちゃんと作ってあれば、.by引数にそれを入れることでもできるわけです。 ただし、実はこの方法も時間がかかる処理で、rowwise()のときと同じくらいの処理時間がかかります。
Code
microbenchmark::microbenchmark(
".by" = {
df_bfi |>
mutate(
score_A = mean(c_across(starts_with("A"))),
score_C = mean(c_across(starts_with("C"))),
score_E = mean(c_across(starts_with("E"))),
score_N = mean(c_across(starts_with("N"))),
score_O = mean(c_across(starts_with("O"))),
.after = q_age,
.by = id
)
},
times = 5L # for saving time
)Unit: seconds
expr min lq mean median uq max neval
.by 4.452133 4.503107 4.554612 4.581422 4.597017 4.63938 5rowMeans(pick(...))
上記の.by引数に突っ込めるrowwise()みたいな関数ってないのかな~と探していたときに、たまたまこんな記事を見つけました。
base::rowMeans()にdplyr::pick()4で列を選択して入れるという技です。pick()はmutate()やdplyr::summrise()のような関数の中でtidy-selectの文法を使ってdataframe列を選択できる関数です。pick()の戻り値がdataframeであること、rowMeans()は引数にdataframeも入れられること、rowMeans()の戻り値は各行の値の平均値を収めたベクトルであることを利用して、rowwise()を使わずに実質的にrowwise処理をしてしまおうというわけですね。
df_bfi |>
mutate(
score_A = rowMeans(pick(starts_with("A"))),
score_C = rowMeans(pick(starts_with("C"))),
score_E = rowMeans(pick(starts_with("E"))),
score_N = rowMeans(pick(starts_with("N"))),
score_O = rowMeans(pick(starts_with("O"))),
.after = q_age
)# A tibble: 2,800 × 34
id q_gender q_education q_age score_A score_C score_E score_N score_O A1 A2 A3 A4 A5
<chr> <int> <int> <int> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <int> <int> <int> <int>
1 61617 1 NA 16 4 2.8 3.8 2.8 3 5 4 3 4 4
2 61618 2 NA 18 4.2 4 5 3.8 4 5 4 5 2 5
3 61620 2 NA 17 3.8 4 4.2 3.6 4.8 2 4 5 4 4
4 61621 2 NA 17 4.6 3 3.6 2.8 3.2 3 4 6 5 5
5 61622 1 NA 17 4 4.4 4.8 3.2 3.6 5 3 3 4 5
6 61623 2 3 21 4.6 5.6 5.6 3 5 1 6 5 6 5
7 61624 1 NA 18 4.6 4.4 4.2 1.4 5.4 5 5 5 3 5
8 61629 1 2 19 2.6 3.4 2.4 4.2 4.2 3 3 1 5 1
9 61630 1 1 19 3.6 4 NA 3.6 5 3 3 6 3 3
10 61633 2 NA 17 5.4 5.6 4.8 4.2 5.2 5 5 6 6 5
# ℹ 2,790 more rows
# ℹ 20 more variables: C1 <int>, C2 <int>, C3 <int>, C4 <dbl>, C5 <dbl>, E1 <dbl>, E2 <dbl>, E3 <int>,
# E4 <int>, E5 <int>, N1 <int>, N2 <int>, N3 <int>, N4 <int>, N5 <int>, O1 <int>, O2 <dbl>, O3 <int>,
# O4 <int>, O5 <dbl>これもまた今までの書き方と同じ結果が返ってきています。mean()がrowMeansに、c_across()がpick()に変わっただけなので、コードの可読性も悪くない気がします。
そして処理速度ですが、上記2つに比べてとても速いです。
Code
microbenchmark::microbenchmark(
"base::rowMeans" = {
df_bfi |>
mutate(
score_A = rowMeans(pick(starts_with("A"))),
score_C = rowMeans(pick(starts_with("C"))),
score_E = rowMeans(pick(starts_with("E"))),
score_N = rowMeans(pick(starts_with("N"))),
score_O = rowMeans(pick(starts_with("O"))),
.after = q_age
)
}
)Unit: milliseconds
expr min lq mean median uq max neval
base::rowMeans 4.3955 4.775 5.446678 5.0946 5.985 9.3616 100上記2つは単位が秒だったのに、こちらの単位はミリ秒です。つまり、4ミリ秒程度で処理が終わっています。
ちなみに、もし各項目の合計得点が尺度得点である場合は、base::rowSums()を使えばいいです。
apply(MARGIN = 1)
そういえば、行での計算はbase::apply(MARGIN = 1)でもできるのを思い出しました。引数には同じくpick()で選んだ列を入れて、関数にmean()を選択すれば同じ結果が得られるはずです。
df_bfi |>
mutate(
score_A = apply(
pick(starts_with("A")),
MARGIN = 1,
FUN = mean
),
score_C = apply(
pick(starts_with("C")),
MARGIN = 1,
FUN = mean
),
score_E = apply(
pick(starts_with("E")),
MARGIN = 1,
FUN = mean
),
score_N = apply(
pick(starts_with("N")),
MARGIN = 1,
FUN = mean
),
score_O = apply(
pick(starts_with("O")),
MARGIN = 1,
FUN = mean
),
.after = q_age
)# A tibble: 2,800 × 34
id q_gender q_education q_age score_A score_C score_E score_N score_O A1 A2 A3 A4 A5
<chr> <int> <int> <int> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <int> <int> <int> <int>
1 61617 1 NA 16 4 2.8 3.8 2.8 3 5 4 3 4 4
2 61618 2 NA 18 4.2 4 5 3.8 4 5 4 5 2 5
3 61620 2 NA 17 3.8 4 4.2 3.6 4.8 2 4 5 4 4
4 61621 2 NA 17 4.6 3 3.6 2.8 3.2 3 4 6 5 5
5 61622 1 NA 17 4 4.4 4.8 3.2 3.6 5 3 3 4 5
6 61623 2 3 21 4.6 5.6 5.6 3 5 1 6 5 6 5
7 61624 1 NA 18 4.6 4.4 4.2 1.4 5.4 5 5 5 3 5
8 61629 1 2 19 2.6 3.4 2.4 4.2 4.2 3 3 1 5 1
9 61630 1 1 19 3.6 4 NA 3.6 5 3 3 6 3 3
10 61633 2 NA 17 5.4 5.6 4.8 4.2 5.2 5 5 6 6 5
# ℹ 2,790 more rows
# ℹ 20 more variables: C1 <int>, C2 <int>, C3 <int>, C4 <dbl>, C5 <dbl>, E1 <dbl>, E2 <dbl>, E3 <int>,
# E4 <int>, E5 <int>, N1 <int>, N2 <int>, N3 <int>, N4 <int>, N5 <int>, O1 <int>, O2 <dbl>, O3 <int>,
# O4 <int>, O5 <dbl>これまでの方法と同じ結果が返ってきました。apply()は引数MARGINに入れるのって0or1だっけ1or2だっけ?どっちが行でどっちが列だっけ?となるので、あんまり使ってないです。(覚えればいいんですけど指定は1or2で、1が行に対して、2が列に対しての計算です。)
処理速度に関してはどうでしょうか。
Code
microbenchmark::microbenchmark(
"apply_margin1" = {
df_bfi |>
mutate(
score_A = apply(
pick(starts_with("A")),
MARGIN = 1,
FUN = mean
),
score_C = apply(
pick(starts_with("C")),
MARGIN = 1,
FUN = mean
),
score_E = apply(
pick(starts_with("E")),
MARGIN = 1,
FUN = mean
),
score_N = apply(
pick(starts_with("N")),
MARGIN = 1,
FUN = mean
),
score_O = apply(
pick(starts_with("O")),
MARGIN = 1,
FUN = mean
),
.after = q_age
)
}
)Unit: milliseconds
expr min lq mean median uq max neval
apply_margin1 69.1538 72.07505 75.1989 73.648 76.3195 178.5305 100bfiデータだと70ミリ秒くらいで済むみたいです。rowMeans()よりは遅いですが、rowwise()と.byよりは速いみたいですね。
Comparison
せっかくなので処理速度を一度に比べてみます。ただし、このままbfiデータでベンチマークするととんでもない時間がかかるので、もっと数が少ないirisデータで比較します。
df_iris <- iris |>
as_tibble() |> # for better printing
rowid_to_column() # for equal results
head(df_iris)# A tibble: 6 × 6
rowid Sepal.Length Sepal.Width Petal.Length Petal.Width Species
<int> <dbl> <dbl> <dbl> <dbl> <fct>
1 1 5.1 3.5 1.4 0.2 setosa
2 2 4.9 3 1.4 0.2 setosa
3 3 4.7 3.2 1.3 0.2 setosa
4 4 4.6 3.1 1.5 0.2 setosa
5 5 5 3.6 1.4 0.2 setosa
6 6 5.4 3.9 1.7 0.4 setosa Code
res <- microbenchmark::microbenchmark(
"rowwise" = {
df_iris |>
rowwise() |>
mutate(
sepal = mean(c_across(starts_with("Sepal"))),
petal = mean(c_across(starts_with("Petal")))
) |>
ungroup()
},
".by" = {
df_iris |>
mutate(
sepal = mean(c_across(starts_with("Sepal"))),
petal = mean(c_across(starts_with("Petal"))),
.by = rowid
)
},
"rowMeans" = {
df_iris |>
mutate(
sepal = rowMeans(pick(starts_with("Sepal"))),
petal = rowMeans(pick(starts_with("Petal")))
)
},
"apply_margin1" = {
df_iris |>
mutate(
sepal = apply(
pick(starts_with("Sepal")),
MARGIN = 1,
FUN = mean
),
petal = apply(
pick(starts_with("Petal")),
MARGIN = 1,
FUN = mean
)
)
},
check = "equal"
)
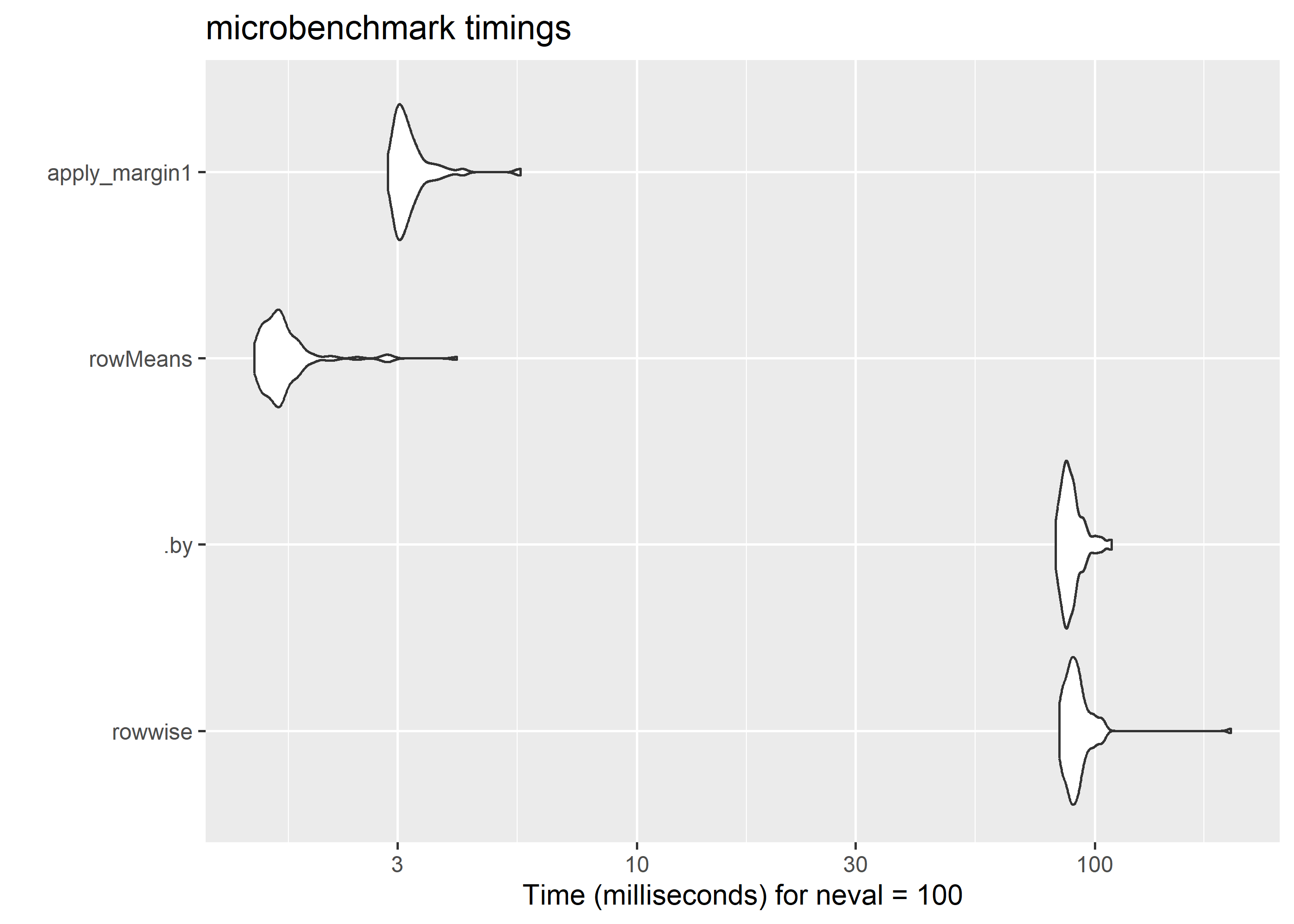
resUnit: milliseconds
expr min lq mean median uq max neval cld
rowwise 83.6199 87.56705 92.048610 90.2374 93.21040 198.1570 100 a
.by 81.9493 85.52320 89.220028 87.6496 90.60810 108.6058 100 b
rowMeans 1.4587 1.56730 1.722859 1.6520 1.73630 4.0320 100 c
apply_margin1 2.8520 2.99130 3.212039 3.0942 3.26395 5.5633 100 c圧倒的にrowMeans()が速いです。桁が違います。元記事の比較データだと、10000行のデータでもrowMeans()を使う方法が圧倒的に速いです。dplyrのarticle(Row-wise operations)にも書いてありましたが、速さを求めるならこっちと言うのもわかります。base関数はやはり侮れません。apply()も行数が少なければ高速っぽいです。
Rfast::rowmeans()
同じノリでRfast::rowmeans()もイケるか…?と思って試してみました。Rfast5パッケージの関数は基本はmatrixかつNAなしじゃないと使えない(内部で使ってるCppの都合でNAがあると良くないとのこと)のですが、Helpを見る限りrowmeans()はdataframeでも計算してくれそうな感じっぽいので試してみました。
Error in `mutate()`:
ℹ In argument: `score_A = Rfast::rowmeans(pick(starts_with("A")))`.
Caused by error:
! Not compatible with requested type: [type=list; target=double].ダメでした。ということで、無理やりmatrixにして再挑戦します。
df_bfi |>
mutate(
score_A = pick(starts_with("A")) |>
as.matrix() |>
Rfast::rowmeans(),
.after = q_age
)# A tibble: 2,800 × 30
id q_gender q_education q_age score_A A1 A2 A3 A4 A5 C1 C2 C3 C4 C5 E1
<chr> <int> <int> <int> <dbl> <dbl> <int> <int> <int> <int> <int> <int> <int> <dbl> <dbl> <dbl>
1 61617 1 NA 16 4 5 4 3 4 4 2 3 3 3 3 4
2 61618 2 NA 18 4.2 5 4 5 2 5 5 4 4 4 3 6
3 61620 2 NA 17 3.8 2 4 5 4 4 4 5 4 5 2 5
4 61621 2 NA 17 4.6 3 4 6 5 5 4 4 3 2 2 2
5 61622 1 NA 17 4 5 3 3 4 5 4 4 5 4 5 5
6 61623 2 3 21 4.6 1 6 5 6 5 6 6 6 6 4 5
7 61624 1 NA 18 4.6 5 5 5 3 5 5 4 4 5 4 3
8 61629 1 2 19 2.6 3 3 1 5 1 3 2 4 5 3 4
9 61630 1 1 19 3.6 3 3 6 3 3 6 6 3 3 2 2
10 61633 2 NA 17 5.4 5 5 6 6 5 6 5 6 5 6 5
# ℹ 2,790 more rows
# ℹ 14 more variables: E2 <dbl>, E3 <int>, E4 <int>, E5 <int>, N1 <int>, N2 <int>, N3 <int>, N4 <int>,
# N5 <int>, O1 <int>, O2 <dbl>, O3 <int>, O4 <int>, O5 <dbl>できました。でも、わざわざmatrixにしなきゃいけいないのはめんどくさいです。
ちなみに、bfiデータの場合、rowMeans()の方法と処理時間には差がつきません。同じくらい速いです。
Code
microbenchmark::microbenchmark(
"base::rowMeans" = {
df_bfi |>
mutate(
score_A = rowMeans(pick(starts_with("A"))),
score_C = rowMeans(pick(starts_with("C"))),
score_E = rowMeans(pick(starts_with("E"))),
score_N = rowMeans(pick(starts_with("N"))),
score_O = rowMeans(pick(starts_with("O"))),
.after = q_age
)
},
"Rfast::rowmeans" = {
df_bfi |>
mutate(
score_A = pick(starts_with("A")) |>
as.matrix() |>
Rfast::rowmeans(),
score_C = pick(starts_with("C")) |>
as.matrix() |>
Rfast::rowmeans(),
score_E = pick(starts_with("E")) |>
as.matrix() |>
Rfast::rowmeans(),
score_N = pick(starts_with("N")) |>
as.matrix() |>
Rfast::rowmeans(),
score_O = pick(starts_with("O")) |>
as.matrix() |>
Rfast::rowmeans(),
.after = q_age
)
}
)Unit: milliseconds
expr min lq mean median uq max neval cld
base::rowMeans 4.2664 4.34500 4.555280 4.4065 4.5439 8.7891 100 a
Rfast::rowmeans 4.2167 4.28615 4.673185 4.3685 4.5172 8.2862 100 aggplot2::diamondsは約54000行あるんですが、計算させてみるとrowMeans()よりも微妙に速かったりします。ただし1ミリ秒も差がつかないので体感できないです。
microbenchmark::microbenchmark(
"base::rowMeans" = {
diamonds |>
mutate(
res = pick(depth:z) |>
rowMeans()
)
},
"Rfast::rowmeans" = {
diamonds |>
mutate(
res = pick(depth:z) |>
as.matrix() |>
Rfast::rowmeans()
)
},
check = "equal"
)Unit: milliseconds
expr min lq mean median uq max neval cld
base::rowMeans 2.2124 2.37665 2.779288 2.51615 2.70125 5.3981 100 a
Rfast::rowmeans 1.5674 1.70840 2.082682 1.83450 2.08915 6.0799 100 bものすごくデータ数が多いときなら、選択肢に入る方法かもしれません。
Conclusion
今回はwideなデータにおいてdplyr::rowwise()を使わないで尺度得点の算出というrowwiseな処理をしてみました。個人的にはbase::row*(pick(...))を使うのもありだと思いました。可読性もそんなに悪くないと思います。一方で、個人ごとに計算しているというのを明示したい場合は、rowwise()を挟んだり.by = ID列の方が分かりやすい気もします。サンプルサイズやコードを共有するか否かなどを考慮して使い分けるのがいいかもしれません。